Sie befinden sich hier
Inhalt
Forschungsschwerpunkt
Die Arbeitsgruppe fokussiert sich auf die molekularbiologische Charakterisierung und bioinformatische Modellierung von Lebererkrankungen. Dies umfasst insbesondere Themen wie Leberfibrose, Lebertumoren, autoimmune Lebererkrankungen und Digitalisierung in der Medizin, mit dem Ziel, Krankheitsmodelle für Prognosen und Medikamentenansprechen aber auch zukunftsweisende Versorgungsmodelle zu entwickeln. MEHR ERFAHREN
Molekularbiologie
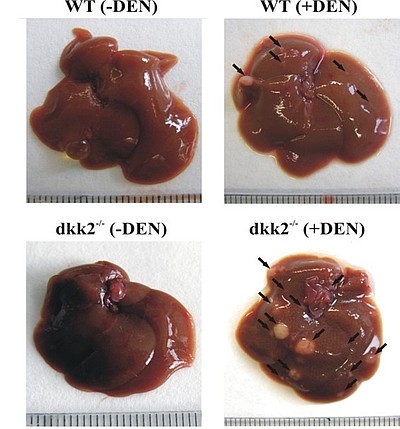
Genveränderte Mausmodelle (z.B. PDGF-B, TGFβ1, Dkk2) zeigen relevante Einflüsse auf Leberfibrose und Karzinogenese. Hierbei werden molekulare Veränderungen auf DNA-, RNA- und Proteinebene untersucht, um ein fibrogenetisches oder tumorbiologisches Gesamtkonzept zu entwickeln. Mikrobiom-Analysen vor und nach bariatrischen Operationen ergänzen die personalisierte Therapie auch auf dem Gebiet der Fettlebererkrankungen. MEHR ERFAHREN
Bioinformatische Modellierung der Krankheitsentstehung und Progression
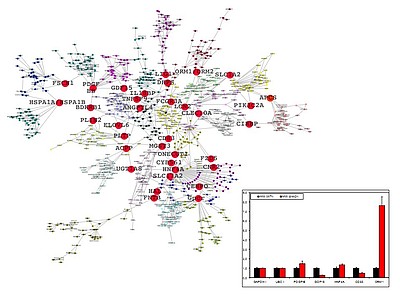
Big Data-Analysen sowie Generierung und Analyse von OMICS-Technologien sind in dr Arbeitsgruppe zur Prognose- und Therapiemodellierung komplexer biologischer Prozesse etabliert. Bioinformatische Ansätze und mehrstufige biologische Modellierung (DNA, RNA, Proteine) werden zur Entschlüsselung von Krankheitsmechanismen genutzt. MEHR ERFAHREN
Internationale Projekte Kooperationen Hepatologie
- China: Einfluss des Mikrobioms auf Lebererkrankungen. Kooperation Prof. Li, Dr. Wong, Dr. Wang, State Key Laboratory for Diagnosis and Treatment of Infectious Diseases, Hangzhou, China. Förderung durch DFG/NSFC.
- Kolumbien: Verbesserung der hepatologischen Versorgung durch digitale Technologien in unterversorgten Regionen, Digital Hepatologie Hub als gemeinsames Akademisches Zentrum Kooperation Prof. Navas, Grupo Gastrohepatología, Facultad de Medicina, Universidad de Antioquia, Kolumbien. Förderung durch Else Kröner-Fresenius Stiftung, Gesellschaft für Internationale Zusammenarbeit. .
- Paraguay: Verbesserung der hepatologischen Versorgung durch digitale Technologien in unterversorgten Regionen. Kooperation Prof. Girala, Universidad Nacional de Asunción, Paraguay. Förderung durch Else Kröner-Fresenius Stiftung, Gesellschaft für Internationale Zusammenarbeit.
- Argentinien: Medikamentöse Beeinflussung der Tumorentwicklung. Kooperation Prof. Ridruejo, Prof. Alvarez, Centro de Educación Médica e Investigaciones Clínicas Norberto Quirno “CEMIC”. Buenos Aires, Argentinien.
Digitale Gesundheitsprojekte
Die Klinische Kooperationseinheit Healthy Metabolism am Zentrum für Präventivmedizin und Digitale Gesundheit (CPD) zielt auf die translationale Übertragung von Grundlagenforschung, insbesondere am ECAS, in die klinische Routine ab. Digitale und telemedizinische Lösungen sollen dabei insbesondere in ländlichen Regionen verfügbar gemacht werden. Der Fokus liegt auf innovativen Versorgungsstrukturen für MASLD mit fünf Kernzielen: KI-gestützte Biomarker-Analyse, personalisierte telemedizinische Betreuung, app-basierte Versorgung, digitale Vernetzung von Patienten und Kliniken über tragbare Sensoren sowie der Aufbau digitaler Versorgungsstrukturen für urbane und ländliche Gebiete. Zur Evaluation innovativer Versorgungsstrukturen bestehen enge Kooperationen u.a. mit Prof. Sniehotta (Public Health), Prof. Hesser (Datenmodellierung) und Dr. Siegel (Medizininformatik) and der medizinischen Fakultät Mannheim. Gefördert wird das Projekt durch das Ministerium für Wissenschaft und Kunst Baden-Württemberg. MEHR ERFAHREN
Evaluation innovativer Versorgungsstrukturen (z.B. sensorbasierte Vernetzung bei MASLD). Kooperation Prof. Sniehotta, PublicHealth, Prof. Hesser, komplexe Datenmodellierung, Dr. Siegel, Medizininformatik, Medizinische Fakultät Mannheim. Förderung durch Ministerium für Wissenschaft und Kunst, Baden-Württemberg.
Einwerben und Aufbau des Zentrums für Digitale Früherkennung und Prävention (ZDFP) mit KI-basierten Methoden zur Reduktion von Volkskrankheiten.
Use Case Fatty Liver Disease im BMBF-LeMeDaRT Konsortium zur Optimierung von Datenbereitstellung im Gesundheitssystem. Kooperation Prof. Sniehotta, PublicHealth, Prof. Hesser, komplexe Datenmodellierung, Dr. Siegel, Medizininformatik, Medizinische Fakultät Mannheim. Förderung durch BMBF.
BW-ZDFP: Video
Ziel: Kombination molekularer Erkenntnisse mit künstlicher Intelligenz und digitaler Infrastruktur zur Verbesserung der Diagnose, Therapie und Prävention von Lebererkrankungen sowie zur Etablierung zukunftsweisender Versorgungsmodelle.
Kontextspalte
Ansprechpartner

Prof. Dr. med. Dr. rer. nat. Andreas Teufel
Sektionsleiter
Phone +49 621/383-6676
Fax +49 621/383-3805
Doktor- und Masterarbeiten
Bei Interesse für eine Master- oder Doktorarbeit wenden Sie sich bitte an Prof. Dr. med. Dr. rer. nat. Andreas Teufel
Publikationen
Publikationen der Arbeitsgruppe finden Sie unter pubmed.
Drittmittelförderung
- Sino-German Center for Research Promotion (SGC); Deutsche Forschungsgemeinschaft (DFG) National Natural Science Foundation of China (NSFC).
- Forum Gesundheitsstandort Baden-Württemberg
- Alexander von Humboldt Stiftung, Bonn
- Stiftung Biomedizinische Alkoholforschung
- Gilead Sciences Inc., Foster City, CA, USA
- IPSEN Pharma GmbH, München
- Bundesministerium für Bildung und Forschung (BMBF)
- Gesellschaft für Internationale Zusammenarbeit
- Else Kröner-Fresenius-Stiftung